

ویروس کرونا 2019 (COVID-19) ناشی از سندرم تنفسی حاد کرونا ویروس 2 (SARS-CoV-2) همچنان تأثیر فوقالعادهای بر روی بیماران و دستگاههای مراقبتهای بهداشتی در سراسر جهان دارد. در مبارزه با این بیماری جدید، برای اطمینان از قرنطینه بهموقع و درمان، نیاز جدی به ابزار غربالگری سریع و مؤثر برای شناسایی بیماران آلوده به COVID-19 وجود دارد.
در حال حاضر، آزمایش RT-PCR روش اصلی غربالگری COVID-19 است، زیرا میتواند اسید ریبونوکلئیک SARS-CoV-2 (RNA) را در نمونههای خلط جمع شده از دستگاه تنفسی فوقانی تشخیص دهد. آزمایش RT-PCR یک فرآیند زمانبر است که در حال حاضر تقاضای زیادی برای آن وجود دارد. این امر منجر به تأخیرهای احتمالی در دستیابی به نتایج آزمون میشود.
تصویربرداری توموگرافی از قفسه سینه (CT) به دلیل حساسیت بالای آن، بهعنوان یک ابزار غربالگری جایگزین برای عفونت COVID-19 پیشنهاد شده است و ممکن است در صورت استفاده بهعنوان مکمل آزمایش RT-PCR، مؤثر باشد. تصویربرداری CT در مراحل اولیه بیماری همهگیر COVID-19، بهویژه در آسیا، استفاده گستردهای را به دنبال داشت. [1]
توموگرافی رایانهای (CT) از پرتوهای اشعه ایکس برای به دست آوردن شدت پیکسلهای سهبعدی در بدن انسان استفاده میکند. کاتد گرم شده پرتوهای پرانرژی (الکترون) آزاد میکند که این خود باعث آزاد شدن انرژی به شکل تابش اشعه X میشود. اشعه ایکس از بافتهای بدن انسان عبور کرده و به یک ردیاب در طرف دیگر برخورد میکند. یک بافت متراکم (استخوانها) نسبت به بافتهای نرم (بهعنوان مثال چربی) اشعه بیشتری جذب میکند. وقتی اشعه ایکس در بدن جذب نمیشود (از ناحیه هوا در داخل ریهها عبور میکند) و به آشکارساز میرسد، آنها را سیاه میبینیم. در مقابل، بافتهای متراکم به رنگ سفید نشان دادهشدهاند. بهاینترتیب، تصویربرداری CT میتواند تفاوتهای تراکم را تشخیص داده و یک تصویر سهبعدی از بدن ایجاد کند. [2]
تصاویر CT-scan که در بیمارستانها گرفته میشود دارای فرمت DICOM هستند که مخفف Digital Imaging and Communications in Medicine است. این فرمت یک فرمت استاندارد جهانی است. فایلهای DICOM علاوه بر تصویر دارای اطلاعات دیگری هستند که به آنها header گفته میشود که در آنها اطلاعات مربوط به بیمار، پروتکلهای تصویربرداری و ... آمده است.
ریه یک اندام سهبعدی است. دستگاه CT-scan از بالای شانهها شروع به تصویربرداری از مقطعهای دوبعدی میکند و به سمت انتهای ریه (نزدیک شکم) حرکت میکند. پس در تصویربرداری از ریهی هر شخص تعدادی فایل DICOM بهدست میآید که مجموع آنها ریهی فرد را نشان میدهد.

با توجه به پروتکلهای محل تصویربرداری، مقاطع تصویربرداری ممکن کم و زیاد شود. هر چه مقاطع بیشتر باشد، تعداد تصاویر بیشتری برای یک شخص ایجاد میشود. همانطور که گفته شد، در قسمت header، اطلاعات تکمیلی وجود دارد که یکی از آنها شماره slice هر تصویر از بین تصاویر یک شخص است. در فرآیندهای آموزش، اگر نیاز باشد ترتیب تصاویر رعایت شود بهترین کار مرتب کردن دادهها بر اساس شماره slice است.
فایلهای DICOM با پسوند .dcm ذخیره میشوند. در python کتابخانههایی برای خواندن تصاویر DICOM وجود دارد که چگونگی کار با برخی از آنها را میتوانید در زیر مشاهده کنید.
import pydicom
ds = pydicom.dcmread(/path/to/dcm/file)
image_2d = ds.pixel_array.astype(float)
from medpy.io import load
image_2d, image_header = load(/path/to/dcm/file)
میزان جذب اشعه X در تصاویر CT-scan در مقیاس Hounsfield اندازهگیری میشود. در این مقیاس، شدت اشعه ثبتشده برای هوا به 1000- و این مقدار برای آب مقطر به صفر نسبت داده میشود. مقادیر برای برخی از بافتها بهصورت زیر است:
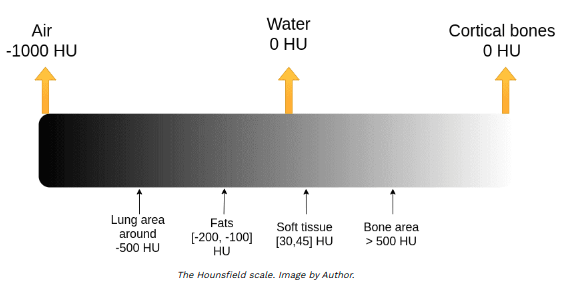
استخوانها از تراکم بالایی برخوردارند، به همین دلیل مقدار اشعهی ثبتشده برای آنها بسیار زیاد است. بیشترین مقادیر متعلق به فلزات است. این اعداد به دلیل وجود نویز ممکن است برای تصاویر واقعی کمی متفاوت باشد.
چشم انسان قادر به دیدن 256 سطح خاکستری قابلتفکیک است، به همین دلیل نمایش تمامی سطوح در مقیاس Hounsfield منطقی نیست. برای حل این مشکل بهصورت استاندارد محدوده خاصی را با توجه به کاربرد در نظر میگیرند. بهصورت قرارداد، در تصاویر پزشکی برای کاهش دامنه Husenfield یک شدت مرکزی، به نام level و یک پنجره، با نام window تعریف میشود. همانطور که در شکل زیر مشاهده میکنید، برای این کار تنها دادههایی که به اندازه window / 2 از level فاصله دارند را مورد بررسی قرار میدهند.
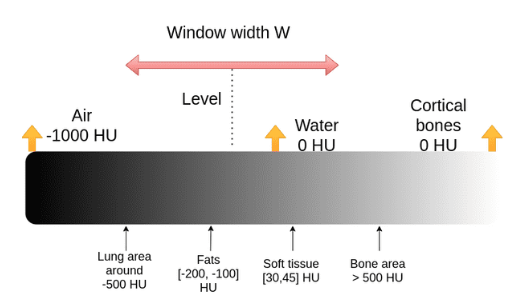
در تصویربرداری از ریه بهصورت استاندارد دو پنجره، متعلق به بافت نرم و بافت ریه در نظر گرفته میشود: [2]
همانطور که گفته شد، بازه تصاویر CT-scan از 1000- تا 2000+ است. کتابخانه pydicom همه این مقادیر را shift میدهد، بهطوری که مقدار کمینه برابر صفر میشود. همچنین تصاویر را به اندازه 90 درجه در جهت ساعتگرد میچرخاند.
کتابخانه medpy مقادیر را در مقیاس Husenfield حفظ میکند. برای این که تصاویر CT-scan به فرم موجود در مجموعهدادهها دربیاید باید با ترانهاده کردن تصویر خواندهشده، چرخش را ایجاد نماییم. در کد زیر نحوه اعمال پنجره مورد نظر و نگاشت و ذخیره سازی آن را به صورت تصویر png میبینیم.
import numpy as np
import png
from medpy.io import load
image_2d, image_header = load(/path/to/dcm/file)
# Selecting the lung window
level = -600
window = 1200
max = level + window/2
min = level - window/2
image_2d[image_2d < min] = min
image_2d[image_2d > max] = max
image_2d = image_2d.astype("float32")
# Mapping to [0, 255]
maxx = image_2d.max()
minn = image_2d.min()
image_2d_scaled = ((image_2d - minn) / (maxx - minn)) * 255.0
# Converting to uint8
image_2d_scaled = np.uint8(image_2d_scaled)
image_2d_scaled = np.squeeze(image_2d_scaled)
image_2d_scaled = np.transpose(image_2d_scaled)
# Writing png
with open(/path/to/png/file, 'wb') as png_file:
w = png.Writer(512, 512, greyscale=True)
w.write(png_file, file2w)
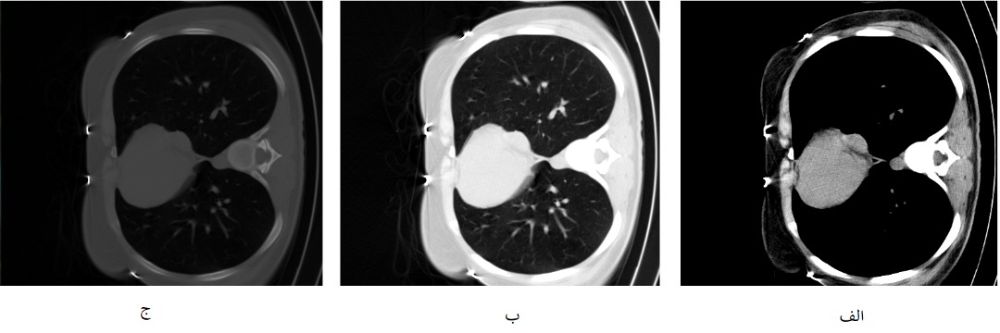
پنجرههای مختلف از یک تصویر در شکل زیر آمده است. تدر این تصویر، (الف) بافت نرم (mediastinum) (ب) بافت ریه (ج) کل بازه میباشد.
بهطورکلی ضایعات ریه به انواع مختلفی تقسیم میشوند که برخی از آنها عبارتند از:
شایعترین یافته غیرعادی ground glass opacities است. ریه در حالت طبیعی باید سیاه و دارای رگههای سفید واضحی باشد که نشاندهنده رگهای خونی در ریه هستند. در صورت درگیری ریه با این نوع ضایعه، بعضی از قسمتهای ریه دارای بخشهای سایهمانند خاکستری میشوند. در عفونتهای شدیدتر، مایعات بیشتری در لوبهای ریه جمع شده، بنابراین بخشهای سایهمانند خاکستری به solid white consolidation تبدیل میشوند. سرانجام، یافتهای به نام crazy paving pattern به دلیل تورم فضای بین بافتی در امتداد دیوارههای ریه به وجود میآید. [3]
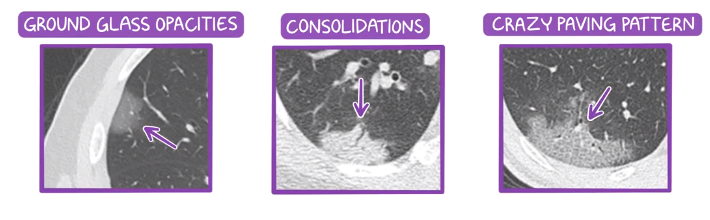
یافتهها نشان میدهد که CT قفسه سینه به ضایعات COVID-19 بسیار حساس است؛ اما solid white consolidation و crazy paving pattern، در بقیه علل ذاتالریه ویروسی مثل آنفولانزا و آدنو ویروس هم دیده میشوند. این ضایعات حتی در انواع مختلف بیماریهای غیرعفونی هم دیده میشوند. [3]
پس از آشنایی با تصاویر CT-Scan میخواهیم به بررسی این مساله در دنیای واقعی بپردازیم. در پروژه واقعی نیاز است که با توجه به سری تصاویر برای هر شخص، تعیین شود که فرد مبتلا به COVID است یا خیر. همچنین مسالههای دیگری از جمله تشخیص میزان پیشرفت بیماری و قطعهبندی ضایعه نیز قابل تعریف است. در ادامه، برخی از مواردی که دانستن آنها به حل مساله کمک میکند را بررسی میکنیم.
واژه NifTi مخفف Neuroimaging Informatics Technology Initiative است که نوعی قالب پرونده برای تصویربرداری عصبی است. پروندههای NifTi بهطورمعمول در انفورماتیک تصویربرداری برای تحقیقات علوم اعصاب و حتی نورورادیولوژی مورد استفاده قرار میگیرند. همانطور که گفته شد فرمت استاندارد مراقبتهای بالینی، پروندههای DICOM هستند. با این حال، برخی از ابزارهای انفورماتیک تصویربرداری میتوانند پروندههای DICOM را بهصورت خودکار به قالب NifTi تبدیل کنند. فایلهای NifTi با پسوند .nii ذخیره میشوند. [4]
همه ما با تصاویر png آشنا هستیم. هر دو فرمت dcm و nii قابل تبدیل به png هستند. برای این کار کافی است که به بازه 0 تا 255 نگاشته شوند. با ترفندهایی میتوان png را نیز به DICOM و NifTi تبدیل کرد ولی این کار با از دست رفتن اطلاعات همراه است که ممکن است جزئیات حائز اهمیتی باشند. همچنین این کار باعث از دست رفتن اطلاعات header نیز میشود.
مجموعه دادههای بسیاری از هر سه فرمت گفته شده در این زمینه وجود دارند که در ادامه به چالشهای کار با هر کدام از آنها میپردازیم:
در اکثر این دادهها نحوهی تبدیل تصاویر از فرمت اصلی و بازه انتخابشده مشخص نیست. این مسئله تکرارپذیری را دشوار میکند. اگر مدلی را بر روی این دادهها آموزش دهیم، برای تست روی دادههای واقعی ابتدا باید فایلهای DICOM را به png تبدیل کنیم که با توجه به نامشخص بودن نحوه تبدیل، دقت افت خواهد کرد.
در این دادهها تصاویر ابتدایی و انتهایی ریه وجود ندارد، یعنی فقط ریههای باز موجود است. در مسائل واقعی و موارد کلینیکی قرار است که از کل تصاویر ریه شخص تصمیم گرفته شود که شخص دارای COVID است یا خیر. اگر مدل با این دادهها آموزش داده شود، چون موارد ریه بسته در training set نیست، در زمان تست واقعی، ریههای بسته توسط مدل اشتباه تشخیص داده میشود و دقت کل مدل افت میکند. برای حل این مشکل، سادهترین راهکار حذف تصاویر ابتدایی و انتهایی ریه در هنگام تست است که البته در حالتهایی که COVID خفیف باشد و صرفاً در slice های ابتدایی و انتهایی، ضایعه دیده شود، باعث افت دقت خواهد شد؛ اما با مطالعات انجامشده احتمال بروز این حالت چندان زیاد نیست.
در این دادهها اکثراً فقط موارد شدید ضایعه COVID وجود دارد. اگر مدل با این دادهها آموزش داده شود، در زمان تست برای موارد خفیف، دقت پایینی خواهد داشت.
در برخی از این دادهها جدا کردن train set ،validation set و test set به درستی انجام نشده است. بهطور مثال slice شماره 10 از یک بیمار در train set و slice شماره 11 در مجموعه دادههای تست قرار دارد که data leakage محسوب میشود و باعث افزایش دقت بهطور کاذب روی این مجموعه داده بهخصوص میشود؛ درحالیکه روی یک test set دیگر بهشدت افت خواهد کرد.
از آنجاییکه فایلهای NifTi هم در مقیاس Husenfield هستند مشکل تبدیل کردن را ندارند. همچنین با توجه به این که این دادهها سهبعدی هستند، کل ریه را شامل میشوند و مشکل حذف ریههای بسته در آنها وجود ندارد. همینطور مشکل جدا کردن train set و test set و یا data leakage هم پیش نمیآید.
بعضی از مجموعه دادهها در این فرمت، میزان شدت بیماری را هم label زدهاند، مثل دیتاست mosmed[5] که case های سالم، case های تا 25٪ درگیر ضایعه، بین 25٪ تا 50٪ درگیر، بین 50٪ تا 75٪ درگیر و بالای 75٪ درگیر را جدا کردهاند. البته این کلاسها imbalanced هستند. از معایب کار با این مجموعه دادهها میتوان به نیازمندی به سختافزار قوی و زمانبر بودن آموزش اشاره کرد.
این مجموعهدادهها، نمونههای جمعآوریشده در بیمارستانها هستند که تمامی فایلهای DICOM هر شخص را در یک folder قرار میدهند.
مشکل این مجموعهدادهها این است که اکثراً folderها برچسبگذاری شدهاند و برچسب هر فایل DICOM مشخص نیست. باید توجه داشت که در مواردی که شخص COVID دارد نیز slice هایی وجود دارد که در آنها ضایعه دیده نمیشود.
در این مطلب با تعریف مساله تشخیص بیماری کرونا از روی تصاویر سیتیاسکن با استفاده از هوش مصنوعی آشنا شدیم. همچنین انواع دادههای موجود برای حل این مساله را بررسی کردیم؛ اما باید توجه داشت که در مسیر حل این مساله چالشهای متفاوتی وجود دارد، مانند این که در مجموعه دادههای عمومی، طبقهبندی با توجه به هر slice صورت میگیرد؛ در صورتی که در دنیای واقعی نیاز است نتیجه برای دنبالهای از تصاویر که به یک شخص تعلق دارند تعیین شود. همچنین تعداد slice هایی که درگیری با کرونا را نشان میدهند، با توجه به مقدار پیشرفت بیماری متفاوت است و تعیین حد آستانه مناسب نیازمند آزمایشهای متعدد است. imbalanced بودن مجموعه دادههای موجود نیز بر پیچیدگیهای مساله اضافه میکند. همچنین باید این نکته را نیز در نظر داشت که در واقعیت طبقهبندی case های دارای ضایعهی خفیف با case های سالم بسیار دشوار است.
منابع
[1] Development and evaluation of an artificial intelligence system for COVID-19 diagnosis
[2] Introduction to medical image processing with Python: CT lung and vessel segmentation without labels
[3] Imaging features of COVID19
[5] MosMedData: Chest CT Scans with COVID-19 Related Findings Dataset
ارسال دیدگاه